模擬計算測試——復達客戶檢測案例
2022年1月2日,本人接到西北某大學同學的計算需求,接到反饋后,第一時間與客戶進行詳細溝通,情況如下:
客戶背景
客戶是西北某大學的一位學生,其想得沒食子酸、兒茶素、金絲桃苷、槲皮苷、積雪草酸、齊墩果酸、熊果酸、金絲桃苷、科羅索酸、α-淀粉酶、α-葡萄糖苷酶、胰脂肪酶、膽固醇脂酶之間的相互作用圖,包括小分子與受體蛋白的結合位點,以什么方式結合在蛋白質的哪一個氨基酸上。能夠得到活性成分與相應酶的結合位點的氨基酸組成,以及結合的方式。
樣品名稱
沒食子酸、兒茶素、金絲桃苷、槲皮苷、積雪草酸分別和α-淀粉酶、α-葡萄糖苷酶
客戶需求
其研究目的如下:
應用分子對接技術,推測單體成分沒食子酸、兒茶素、金絲桃苷、槲皮苷、積雪草酸分別和α-淀粉酶、α-葡萄糖苷酶之間可能的結合位點。
推測單體成分齊墩果酸、熊果酸、金絲桃苷、科羅索酸和胰脂肪酶之間可能的結合位點。
推測單體成分齊墩果酸、熊果酸、科羅索酸和膽固醇脂酶之間可能的結合位點。
解決方案
1.受體結構準備
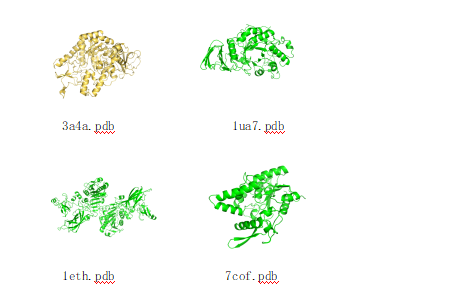
從PDB數據庫中下載3a4a.pdb、1ua7.pdb、1eth.pdb、7cof.pdb結構文件。釀酒酵母α-葡萄糖苷酶PDB:3a4a.pdb,芽孢桿菌α-淀粉酶PDB:1ua7.pdb,豬胰腺-胰脂肪酶PDB:1eth.pdb,假單胞菌膽固醇脂酶PDB:7cof.pdb。用pymol對4個結構文件進行預處理得到初始結構,初始結構用AutoDock Tools 1.5.6 進行處理,計算Gaseigter電荷,并生成pdbqt文件用于分子對接。蛋白結構如下圖:
2.配體結構準備
利用ChemBioDraw生成分子的2D文件,用ChemBio3D生成分子的PDB文件。在ChemBio3D中用MM2方法對小分子進行能量最小化,用于后續的分子對接,采用AutoDock Tools 1.5.6處理配體結構,生成pdbqt文件用于對接。
客戶反饋
客戶對數據結果滿意,無論計算得到的數據還是我們提供的分析報告,以及后續的答疑都非常清晰嚴謹。






 600
600  600
600